|
阅读:9320回复:0
生物信息分析-全外显子组重测序外显子组测序(exome sequencing)是利用芯片或探针杂交富集外显子DNA序列,然后进行高通量测序的方法。由于疾病相关的突变大部分集中在
蛋白编码序列(开放读码框),且外显子只占基因组大小的1%,因此外显子组测序能够、较经济地、针对性地对基因区域进行扫描,鉴定外显子上与疾病相关的碱 基变异。该方法能够获得指定外显子捕获平台探针设计区域及侧翼200bp序列的遗传信息,极大地提高了人类基因组中外显子区域的研究效率,显著降低了研究 成本。 外显子组测序主要用于识别和研究与疾病、种群进化相关的编码区及UTR区域内的结构变异。结合大量的公共数据库
提供的外显子数据,有利于更好地解释所得变异结构之间的关联和致病机理。目前,外显子组测序已广泛用于单基因病(孟德尔疾病)、癌症等复杂疾病的研究,成 为鉴定致病突变、揭示疾病发生机制、提供诊断治疗参考的重要工具。 图片:11.png 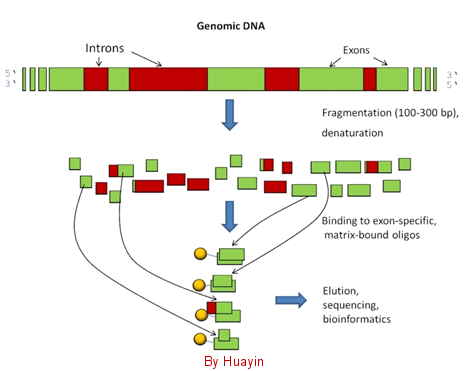
分析流程: 图片:12.png 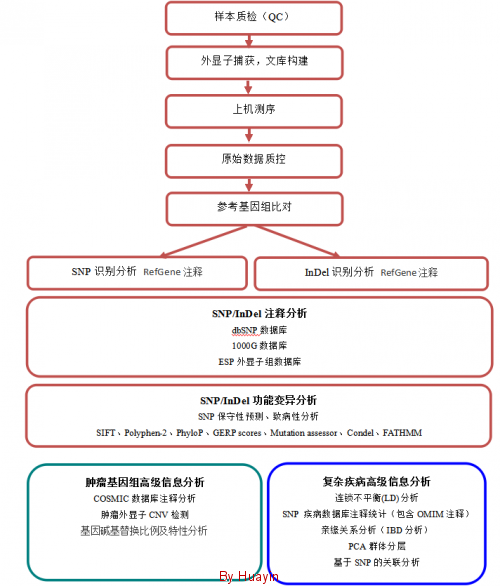
分析目标: 1)
SNP与indel检测 分析模块用于识别重测序数据中的单核苷酸多态性(SNP),本分析采用经验优化的灵敏和准确的分析算法,极大地保证SNP识别的高准确率和重复验证率。数据分析算法的优化考虑多种处理降低7类主要造成假阳性的错误来源(例如PCR错误,低质量区域,依据倍型信息质量得分判断错误等)。 图片:13.png 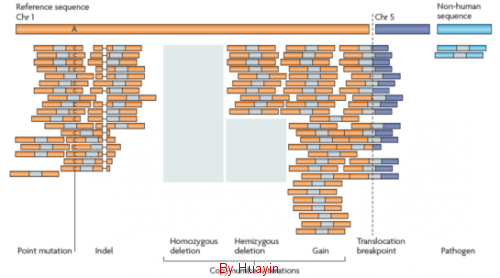
该分析模块采用重新比对优化的处理方式分析,有利于识别测序读长中存在的新Indel位点。提高变异体的识别灵敏度,特异度和识别的效率。对于人类单基因遗传病研究,可以进一步将患病与对照组对应起来,针对插入缺失突变导致的蛋白质编码阅读框的位移分析找出致病位点 2)
体细胞突变分析 本项分析模块主要针对肿瘤基因组相对自身对照的正常细胞基因组的体细胞突变进行系统分析,识别体细胞突变并统计相应体细胞突变类型,包括非同义突变,短插入缺失导致的阅读框移码等。并结合相关背景突变率统计显著的驱动突变基因(即Driver
mutation)。 图片:14.png 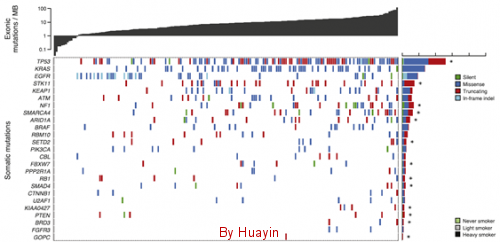
3) 结构变异预测 结构变异(StructureVariation,SV)是基因组变异的一类主要来源,主要由大片段序列(一般>1kb)的拷贝数变异(copy
number variation, CNV)以及非平衡倒位(unbalance inversion)事件构成。SV形成机制包含:(1)同源性介导的直系同源序列区段重组(NAHR);(2)与DNA双链断裂修复或复制叉停顿修复相关的非同源重组(NHR);(3)通过扩展和压缩机制形成可变数量的串联重复序列(VNTR);(4)转座元件插入(一般主要是长/短间隔序列元件LINE/SINE或者伴随TEI相关事件的两者的组合)。本分析模块将识别SNV结构及其断点(breakpoint),统计SNV类型在基因组内的分布状态,并与样本表性数据相关联。 图片:15.png 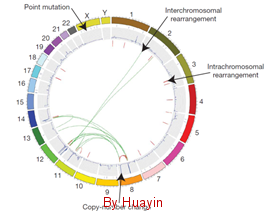
4) 拷贝数变异 本项分析依据深度测序或低覆盖度测序模式,分别可采用不同分析技术准确识别基因组拷贝数变异-CNV。精细度取决于测序深度。测序深度(coverage)与测序数据量直接相关,并与CNV的探测解析度(windows
size)有一定的相关性。另外,通过CNV分析进一步可分析得到杂合性缺失(LOH),用于判断遗传结构变异和变异等位基因在样本中的比例。全基因组CNV分析对于外周血DNA或游离核酸ctDNA,肿瘤组织等样本适用。对于外显子组测序也可进行CNV项目的分析。 b)
目标区域扩增子测序分析 扩增子测序是对特定长度的PCR产物或者捕获的片段进行测序,分析序列中的变异。扩增子测序能够根据不同需求,对目标区域进行高覆盖度的测序,还可以检测到低频突变。技术优势:更高通量,适用于大量样本的特定基因组区域研究。PCR扩增,使用多组PCR引物对目标区域进行扩增,而非探针捕获。高信息度,针对感兴趣的基因组区域进行遗传变异位点的寻找,对特定区域进行深入研究。缩短研究周期:加快文章发表与加速临床应用。节约成本,相比于全基因组重测序,更加经济、高效。 |
|
